Électrophorèse
d’ADN sur gel d’agarose
Introduction
Les multiples applications scientifiques et technologiques impliquant
l'étude de l'ADN font appel à différentes techniques.
Parmi elles, l’électrophorèse sur gel d’agarose ou de polyacrylamide
est une des plus communément utilisées car elle permet de
séparer des molécules en fonction de leur taille, préalable
indispensable dans de multiples applications, notamment pour identifier
des fragments d’ADN découpés par des enzymes, pour identifier
un gène ou pour établir des empreintes génétiques
par Southern Blot.
L’électrophorèse d’ADN sur gel d’agarose ne présente
guère de difficultés techniques et peut être utilisée
aisément en travaux pratiques tant à l’université
qu’au lycée pour familiariser les étudiants avec cette technique
essentielle. En conjonction avec divers outils informatiques désormais
communs (traitement de texte, tableur, logiciels d'acquisition et de traitement
d’images), l’analyse des résultats peut conduire à des développements
importants permettant de mieux comprendre l’intérêt tant scientifique
que pratique des technologies de l’ADN. Différentes possibilités
mettant à profit ces divers outils sont présentées
ci-dessous.
Noter que pour racourcir le temps de chargement de la page, la plupart
des images sont présentées dans un format réduit.
Pour obtenir les images grand format, il faut cliquer sur l'image avec
le bouton gauche de la souris.
-
Mise en pratique de l’électrophorèse
d’ADN sur gel d’agarose.
1- Mélanger tampon TBE et agarose à raison de 0,8 g d'agarose
pour 100 mL de tampon (la proportion d’agarose dépend de la taille
des molécules d’ADN à séparer).
2- Faire fondre l'agarose au four à micro-ondes en surveillant
pour éviter les projections ou au bain marie. Agiter de temps à
autre pour homogénéiser le mélange.
3- Laisser refroidir jusqu'à ce qu’il devienne possible de saisir
le flacon à main nue (environ 60 °C).
4- Placer les joints fournis avec la cuve pour fermer le support de
gel et positionner le peigne à 1 mm du fond et à environ
1 cm de l’extrémité du support. Régler le niveau pour
que le support de gel soit horizontal.
Certains peignes sont réglables avec une vis : il y a alors
une cale pour régler leur hauteur. D’autres sont préadaptés
au support et ne nécessitent pas de cale.
5- Couler lentement le gel sur 3 à 5 mm d'épaisseur en
veillant à ce qu’il entoure bien les dents du peigne.
6- Laisser refroidir, enlever le peigne et les joints. Le gel est prêt
pour le dépôt des échantillons.
NB On peut préparer du gel à l’avance et le conserver
solide au réfrigérateur dans un flacon en verre (rempli aux
2/3 maximum). Au moment de l’emploi, refondre le gel au four à micro-ondes
(ouvrir le bouchon et surveiller pour éviter les projections) ou
au bain-marie bouillant (ouvrir le bouchon). |

Gel d'agarose
(0,8 g/100 mL)
|

Mise en place du peigne et des joints de coulage
(minicuve pour gel 8 x 6,5 cm)
|
-
Préparation et dépôt des échantillons
Pour se familiariser avec cette technique, le plus simple est d’acheter
dans le commerce des échantillons d’ADN, entier ou digéré
par des enzymes de restriction. Les fournisseurs proposent
toute une gamme d’ADN de diverses sources digéré ou non par
des enzymes.
Les échantillons d'ADN peuvent être obtenus en solution
dans un tampon TE (Tris EDTA) ou lyophilisés. Dans ce cas, il sont
remis en solution dans du tampon TE.
Les solutions d'ADN peuvent être conservées indéfiniment
à - 20 °C.
Les échantillons d’ADN sont le plus souvent distribués
par les fournisseurs avec du colorant de charge qu’il suffit de mélanger
aux échantillons avant le dépôt en suivant les proportions
indiquées par le fabricant. Une pipette de précision est
nécessaire pour la suite des opérations.
Protocole
Placer les échantillons d’ADN pendant 3 min à 60-65°C.
Les transférer sur de la glace pendant 2-3 min.
Ces deux opérations sont destinées à empêcher
la ligation de fragments possédant éventuellement des extrémités
cohésives.
Pour le remplissage de puits de 10-12 mm de large bien adaptés
à un usage pédagogique, une proportion de 5 µL de colorant
de charge pour 10 à 15 µL d’ADN convient (de façon
à obtenir de 1 à 4 µg d’ADN par puits).
-
Mélanger le colorant de charge et l’ADN sur un morceau de parafilm
et prélever le mélange avec une micropipette réglée
sur le volume approprié en changeant de cône à chaque
prélèvement.
-
Remplir les puits en faisant attention de ne pas déchirer le fond
du gel avec la pointe de la pipette.
-
Placer le support avec le gel chargé dans la cuve d'électrophorèse
en positionnant les puits du côté de la cathode (pôle
noir).
-
Remplir la cuve de tampon TBE (réutilisable plusieurs fois) en versant
délicatement et très lentement lorsque le gel commence à
être recouvert pour éviter les fuites d’ADN vers le tampon.
-
Fermer la cuve, brancher les fils et mettre sous tension.
-
Laisser migrer jusqu’à ce que le colorant de charge arrive à
proximité du bord du gel (environ 55 min à 100 V pour un
gel de 80 mm dans une minicuve).
-
Couper l’alimentation, débrancher les connections et récupérer
le gel dans son support.
Attention au transport : le gel glisse très facilement de son
support. |
|

Electrophorèse d'ADN sur gel d'agarose 15 x 10 cm (15
puits)
Noter la migration du colorant de charge de la cathode vers l'anode
|
-
Révélation des bandes d’ADN
Attention, le gel doit être manipulé avec soin car il est
très fragile.
-
Recouvrir le gel avec le colorant.
-
Laisser agir de 1 h à une nuit puis vider le colorant (qui peut
être réutilisé). Les bandes d’ADN apparaissent en bleu.
Les gels sont observables à ce stade mais on peut vouloir décolorer
le fond pour améliorer la qualité des photographies.
-
Rincer dans l’eau distillée pendant quelques heures en changeant
l’eau de temps en temps.
-
Photographier le gel avec un appareil numérique ou le scanner.
Les clichés ci-dessous, réalisés avec un scanner,
présentent les résultats obtenus pour deux tailles de gels
d'agarose.
Noter que le contraste des images a été amélioré
par traitement numérique des images afin d'éviter la perte
d'informations résultant de la prise de vue. En effet, les bandes
les moins marquées mais néanmoins visibles à l'oeil
nu ne sont parfois pas détectées par l'appareil de prise
de vue.
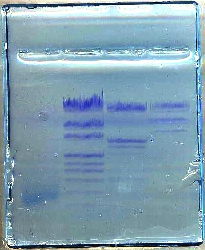
Minigel 8 x 6,5 cm coloré
par le bleu de Nile
|
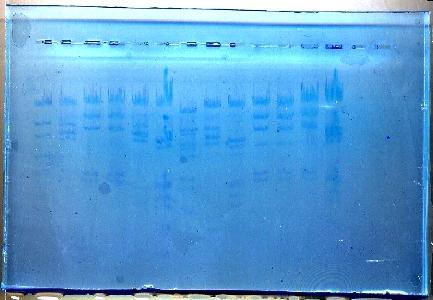
Gel 15 x 10 cm coloré par le bleu de Nile
|
Pistes du minigel :
1 :
ADN génomique
de lentille |
2 :
Marqueurs de taille |
3 :
ADN de phage
lambda digéré par EcoRI et HindIII |
4 :
ADN de phage
lambda digéré par HindIII |
Pistes du gel 15 x 10 :
(gel destiné à évaluer la qualité de l'ADN
conservé au laboratoire depuis parfois plusieurs années avec
décongélations - recongélations successives)
1 :
P
pUC
Mix |
2 :
P
EcoRI |
3 :
P
HindIII |
4 :
S
HindIII |
5 :
P
EcoRI |
6 :
P
EcoRI
+ HindIII |
7 :
S
BstII |
8 :
S
EcoRI |
9 :
P
EcoRI
+ HindIII |
10 :
P
HindIII |
11 :
P
HindIII |
12 :
P
HindIII |
13 :
P
EcoRI
+ HindIII |
Puits 1 : marqueurs de taille : pUC mix (Promega)
Puits 2 à 13 : ADN de phage lambda digéré par
diverses enzymes de restriction. Origine de l'ADN : Promega : P ; Sigma
: S |
. |
-
Traitement des images et détection
de bandes invisibles à l'oeil nu
On peut améliorer la visibilité des bandes, notamment des
moins marquées par le colorant, par des traitements numériques
des images réalisés avec un logiciel de traitement d'images
(PhotoEditor, Paint Shop Pro, Photoshop, etc.). On peut jouer sur
la luminosité, sur le contraste, sur l'intensité relative
des couleurs. On peut également obtenir une image en négatif
souvent plus lisible comme ci-dessous. |
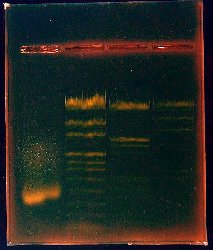
Minigel
|
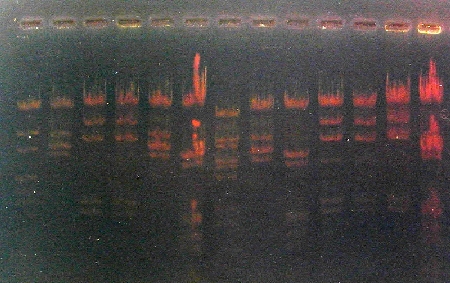
Midigel
|
-
Détection de bandes invisibles par traitement numérique
Un logiciel de traitement d'images numériques donne la possibilité
de mesurer l'intensité lumineuse de chaque pixel de l'image et celle
de ses trois composantes colorées. La représentation graphique
de chacun de ces tracés à l'aide d'un logiciel tableur comme
Excel permet alors de détecter des bandes invisibles à l'oeil
nu en repérant les variations d'intensité sur les courbes.
La figure ci-dessous présente les tracés obtenus avec la
bande 2 du minigel ci-dessus (marqueurs de longueur,
pUCMix). Le logiciel permettant ce type de traitement, développé
par M. Borie, professeur de SVT, est fourni gracieusement par l'auteur.
Vous pouvez l'obtenir en lui envoyant
un courrier.
Un autre logiciel, "Mesurim", développé par J.F. Madre
et librement téléchargeable sur le site
de l'Académie d'Amiens permet également ce type de traitement.
On se référera aux rubriques d'aide disponibles sur ce site
pour son utilisation. |
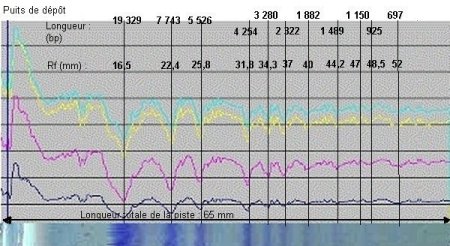
Profils densitométriques (densité et couleurs RVB)
superposés à la piste correspondante du gel
(Piste 1 : marqueurs de taille de l'ADN [pUC Mix, Promega])
Noter que 9 bandes seulement sont visibles à l'oeil nu mais que
les variations simultanées des quatre tracés permettent d'identifier
deux bandes supplémentaires (925 et 697 paires de bases respectivement).
Les bandes étant identifiées, leur distance de migration
est mesurée. Pour cela, l'image obtenue est affichée dans
un logiciel de traitement d'image (PhotoEditor, Paint Shop Pro, Photoshop,
etc.) et la distance de chaque bande au puits est mesurée directement
avec la souris, le logiciel affichant le nombre de pixels correspondant.
Les valeurs obtenues ont été reportées sur le graphique
ci-dessus (Rf en mm).
Connaissant la taille en paires de bases des fragments d'ADN donnée
par le fournisseur, il est alors possible de construire la courbe exprimant
la distance de migration en fonction de la taille des fragments d'ADN.
|
-
Construction de la courbe Distance
de migration = f (taille de l'ADN)
La construction de la courbe nécessite, d'une part, de disposer
des informations données par le fournisseur sur la taille des différents
fragments de restriction présents dans le mélange et, d'autre
part, des mesures réalisées précédemment sur
l'image pour déterminer la distance de chaque bande à l'origine,
c'est à dire au puits de dépôt.
N'importe quel tableur peut être utilisé pour entrer les
données (taille des fragments en abscisses, distances de migration
en ordonnées) et construire le graphique correspondant (Rf =
a(1/log longueur) + b) qui constitue la courbe étalon permettant
ensuite de déterminer la taille de fragments inconnus à partir
de leur distance de migration. En outre, la construction de la courbe permet
de vérifier la bonne adéquation des résultats obtenus
avec la dimension des fragments de restriction indiquée par le fournisseur.
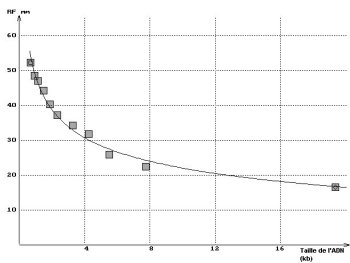
Sur le graphique ci-dessous à gauche on a superposé ainsi
les résultats obtenus avec la piste 2 du minigel
ci-dessus et la courbe théorique. Le tracé peut être
aussi linéarisé en utilisant en abscisses le logarithme de
la taille des fragments d'ADN comme le montre le graphique ci-dessous à
droite ou en utilisant une échelle semi logarithmique.
|
|
 |
Distance de migration en fonction
de la taille des fragments d'ADN |
Distance de migration en fonction
du logarithme de la taille des fragments d'ADN |
-
Détermination de la longueur
de fragments d'ADN de taille inconnue
-
Les tracés ci-dessus peuvent être utilisés pour déterminer
la taille de fragments d'ADN inconnus séparés par électrophorèse.
Pour cela, la distance de migration des bandes de taille inconnue est mesurée
sur le graphique. On a procédé de cette façon pour
la piste 3 du minigel ci-dessus (ADN de phage lambda
digéré entièrement par EcoRI et HindIII) après
avoir identifié par analyse de l'image de la piste les bandes invisibles
à l'oeil nu comme indiqué précédemment. La
figure ci-dessous présente les résultats de cette opération.
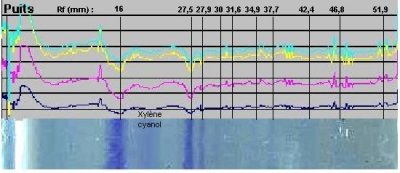
-
Les distances de migration relevées ont été ensuite
utilisées pour déterminer graphiquement la taille prévisible
des fragments en lisant sur la courbe étalon la valeur des abscisses
correspondantes.
La comparaison des valeurs déduites de cette manière
avec les indications du fournisseur permet d'évaluer la qualité
de la séparation électrophorétique réalisée.
Le graphique ci-dessous présente les résultats obtenus.
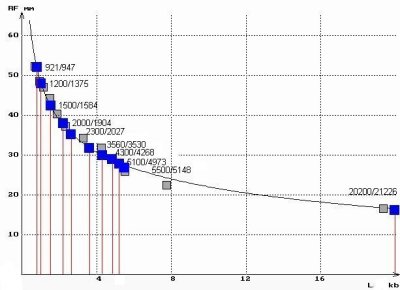
Détermination graphique de la taille de fragments de restriction
Les deux valeurs indiquées pour chaque point correspondent respectivement
à la valeur déterminée graphiquement et à la
valeur théorique attendue pour chaque bande (puisqu'il s'agit ici
d'un échantillon d'ADN dont les produits de digestion sont connus).
|
-
Localisation des sites de restriction
et prévision des résultats
|
-
Les séquences d'ADN conservées dans les banques
de données internationales (EMBL,
Genbank,
etc.) sont accessibles directement à partir des serveurs Web où
elles peuvent être téléchargées aisément.
Pour les exemples donnés ici, la séquence d'ADN du bactériophage
lambda est téléchargeable directement à partir de
cette page (au format html ou au format texte .txt utilisable directement
avec un traitement de texte) en cliquant sur le lien correspondant.
Le génome du phage lambda comporte 48 502 paires de bases et
a une masse moléculaire de 31,5 x 106 daltons.
-
Une fois la séquence chargée, on peut entreprendre de localiser
les sites de restriction dans le génome du phage lambda dont l'ADN
a été utilisé pour réaliser l'électrophorèse.
L'enzyme EcoRI reconnaît la séquence :
..GAATTC..
..CTTAAG..
L'enzyme HindIII reconnaît la séquence :
..AAGCTT..
..TTCGAA..
La fonction rechercher de tout traitement de texte ou du navigateur
permet de localiser les sites de restriction si on lui indique la séquence
à retrouver. Noter que la séquence ne doit pas comporter
d'espaces entre les nucléotides pour que la fonction "rechercher"
puisse trouver tous les sites de restriction. Dans ces conditions, la recherche
de la séquence GAATTC conduit à identifier cinq sites de
restriction situés respectivement à 21226, 26104, 31747,
39168, 44972 générant six fragments. De la même façon,
la recherche de la séquence AAGCTT conduit à identifier six
sites de restriction situés respectivement à 23130, 25157,27479,
36895, 37459, 44141, générant sept fragments. Il est intéressant
d'essayer de prévoir le nombre de fragments produits théoriquement
par l'action simultanée des deux enzymes et de le comparer au nombre
de fragments obtenus lors de l'électrophorèse.
|
-
Solutions
-
Tampon TBE (Tris borate EDTA) pH 8,3
Pour 1000 mL d’eau distillée :
Tris.HCl [tris(hydroxyméthyl)aminométhane] 90 mmol.L-1
: 10,89 g
Acide borique 90 mmol.L-1 : 5,56 g
EDTA 2 mmol.L-1 : 0,74 g (sel disodique de l’acide éthylène
diamine tétraacétique)
-
Tampon TE (Tris EDTA) pH 7,6
Pour 100 mL d’eau distillée :
Tris.HCl [tris(hydroxyméthyl)aminométhane] 1 mol.L-1
: 12,1 g
EDTA (sel disodique de l’acide éthylène diamine tétraacétique)
0,1 mol.L-1 : 3,72 g
-
Colorant de charge pH 8
Pour 10 mL d’eau distillée :
Bleu de bromophénol 3 mmol.L-1 : 0,2 g
Saccharose 1,5 mol.L-1 : 5,1 g
Tris. HCl 10 mmol.L-1 : 0,01 g
-
Solution de coloration pour les gels
-
Solution stock (concentrée 100 fois) à conserver au
réfrigérateur :
Dissoudre 10 mg de bleu de Nile dans 5 mL d’eau distillée.
-
Solution de coloration :
1 mL de la solution stock pour 100 mL d’eau distillée.
Selon la taille de la cuve de coloration, préparer 200 à
300 mL de la solution de coloration.

Cuves à électrophorèse :
Pierron, Bio-Rad,
Polylabo,
Bioblock
Produits chimiques :
SIGMA. L'Isle d'Abeau
Chesnes BP 701 - 38297 Saint Quentin Fallavier
ADN :
EUROMEDEX. BP 80 - 67460 Souffelweyersheim
Séquences d'ADN :
EMBL, Genbank
(Pour le génome du phage lambda, numéros d'accession : M17233,
M24325, V00636) |